
Revolution im HLA-Labor
Bei einer Typisierung werden die für hämatopoetische Stammzelltransplantationen relevanten DNA-Abschnitte des humanen Leukozytenantigen-Systems (HLA) sowie einige weitere Genorte sequenziert, d.h. die Basenabfolge der DNA-Moleküle wird bestimmt. Diese Genorte werden zwischen dem Empfänger und potenziellen Spendern eines Transplantats abgeglichen, um Abstoßungsreaktionen zu vermeiden.
In den vergangenen Jahren hatte das Labor das stetig wachsende Spenderaufkommen der DKMS zu bewältigen und ist Anfang 2013 als eines der ersten Labore dieser Art auf die damals noch revolutionäre und extrem leistungsfähige Methode des Next Generation Sequencing (NGS) im Hochdurchsatz umgestiegen. Während die zuvor verwendeten Kapillar-Sequenzierer in einem Gerätelauf von etwa 40 Minuten 96 DNA-Sequenzen parallel erzeugen konnten und dabei durchschnittliche Leselängen von etwa 800 Basenpaaren erreichten, generieren die derzeit bei uns verwendeten hochparallelen HiSeq 2500-Sequenzierer bis zu 600 Millionen Sequenzstücke (sogenannte Reads) von je 2×250 Basenpaaren Länge. Ein einzelner 2,5-tägiger Gerätelauf eines HiSeq-Instruments erlaubt es damit, über 5000 Spender gleichzeitig zu typisieren.

Im Juni 2016 wurde – dreieinhalb Jahre nach Einführung von NGS im Routinebetrieb – der 2,7-millionste DKMS-Spender mit dem neuen NGS-Verfahren typisiert. Wir haben dies zum Anlass genommen, die im Laufe der Zeit gesammelten Erfahrungen und gewonnenen Erkenntnisse systematisch aufzuarbeiten und konnten diese nun in einem Übersichtsartikel in der renommierten internationalen Fachzeitschrift BMC Genomics publizieren. Der Artikel richtet sich in erster Linie an Molekularbiologen, die ähnliche Methoden für eine Vielzahl biologischer Fragestellungen anwenden wollen, verdeutlicht aber auch die Leistungsfähigkeit der neuen Sequenziermethode. So konnte die Typisierungskapazität des Labors über die Jahre kontinuierlich gesteigert werden. Während 2013 im Monatsmittel noch etwa 30.000 Spender bearbeitet wurden, stieg diese Zahl in den Jahren 2014 und 2015 auf 70.000 Spender, und im Jahr 2016 sogar auf über 110.000 Spender pro Monat.
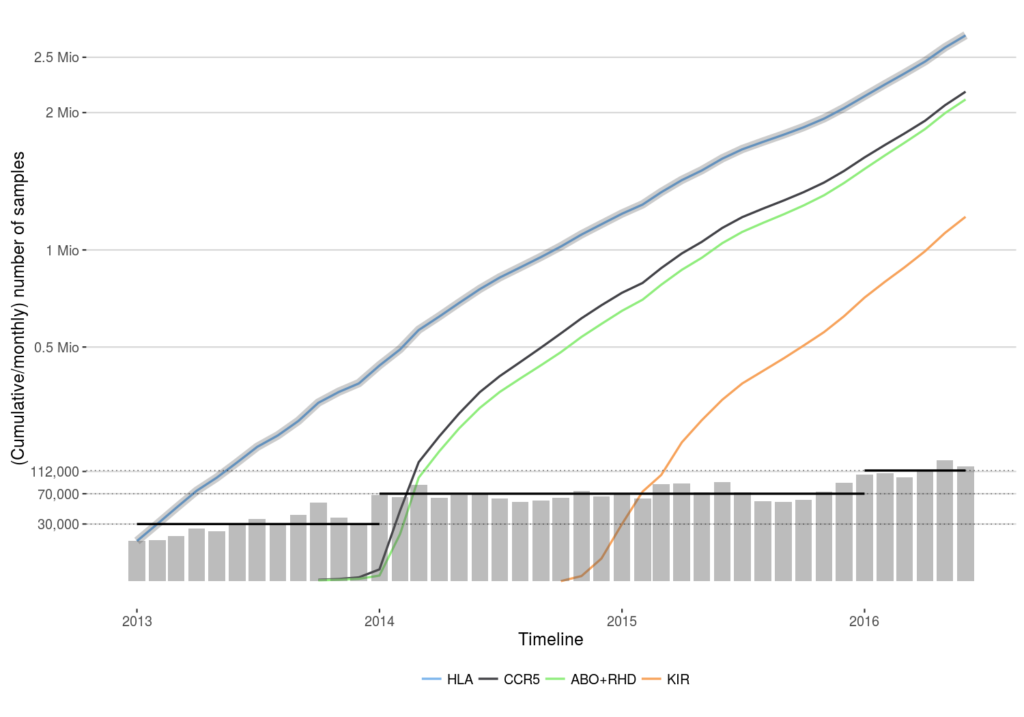
Gleichzeitig wurde das Typisierungsprofil des Labors kontinuierlich erweitert. Wurden zunächst ausschließlich die sechs „klassischen“ HLA-Gene HLA-A, -B, -C, -DQB1, -DRB1 und -DPB1 typisiert, zeigte sich rasch, dass bei geringem zusätzlichen Aufwand weitere potenziell transplantationsrelevante Faktoren ins Profil mit eingeschlossen werden konnten. So werden seit Anfang 2014 bei allen Neuspendern die Blutgruppen und der Rhesusfaktor sowie das Gen CCR5 mitbestimmt. Anfang 2015 wurde das Profil nochmals um die komplexe Gen-Familie KIR erweitert (Update: Seit 2022 wird die Bestimmung der KIR-Gene nur noch bei 10 Prozent aller Neuaufnahmen durchgeführt).
Möglich wurde dies einerseits durch kontinuierliche Neuerungen an der Sequenzier-Technologie und -chemie, andererseits aber auch durch die Optimierung der Laborabläufe, der Automatisierung und einer effizienteren Ausnutzung der erzielten Sequenzierleistung. So wurden anfangs beispielsweise ein gutes Dutzend der sogenannten MiSeq-Geräte – die „kleinen Brüder“ der HiSeqs – zur Sequenzierung eingesetzt. Denn nur MiSeqs konnten zunächst die für unseren Anwendungszweck unabdingbare Leselänge von 2×250 Basenpaaren erreichen. Allerdings sind diese Geräte bei einer Sequenzierleistung von maximal 25 Millionen Reads pro Gerätelauf deutlich kleiner dimensioniert als die jetzt eingesetzten HiSeqs. Mitte 2015 erreichten dann auch die HiSeq-Instrumente diese Leselänge und erst der Umstieg auf HiSeqs schuf die notwendigen Kapazitäten, die KIR Gen-Familie ins Typisierungsprofil aufzunehmen.

Als Beispiel für eine effizientere Ausnutzung der Sequenzierleistung sei der Ausgleich von schwankenden Sequenziertiefen bei verschiedenen Spendern genannt. Dazu muss man wissen, dass beim NGS nicht nur eine Sequenz pro Spender pro Genort erzeugt wird, sondern dass wir mindestens eine 100fache Abdeckung (100fache Sequenziertiefe) jedes Genorts anstreben. Da eine Einzelsequenz mit einer gewissen Wahrscheinlichkeit zufällige Fehler aufweisen kann, dient diese Redundanz zur bioinformatischen Berechnung sogenannter Konsensus-Sequenzen, die dann mit einer hohen Wahrscheinlichkeit der wahren Sequenz entsprechen.
Weil die letztlich erreichte Sequenziertiefe jedoch von einer Vielzahl schwer zu kontrollierender molekularer Faktoren abhängt, muss man oft eine wesentlich höhere mittlere Sequenziertiefe anstreben als theoretisch notwendig wäre, um auch bei „schlecht laufenden“ Proben noch über eine ausreichende Menge an Sequenzen für eine zuverlässige Typisierung zu verfügen. Aufgrund der Masse an Daten, die wir generieren, konnten wir aber nach einiger Zeit einen statistischen Zusammenhang (Michaelis-Menten Sättigungsfunktion) zwischen der Ausgangs-DNA-Konzentration einer Probe und der erreichten Sequenziertiefe beobachten. Diesen Zusammenhang können wir nun verwenden, um Proben über einen weiten Bereich von DNA-Konzentrationen zu equilibrieren und damit die vorhandenen Sequenzier-Ressourcen effizienter zu nutzen.

Grundsätzlich lässt sich sagen, dass die Entwicklung und der Einsatz von Next-Generation Sequenzier-Plattformen die Typisierung von Stammzellspendern revolutioniert hat. Die verfügbare Technologie erlaubt durch eine immense Parallelisierung des Sequenzier-Prozesses eine noch nie dagewesene Erhöhung der Typisierungskapazität bei gleichzeitiger Ausweitung des Typisierungsprofils und der Senkung der Typisierungskosten pro Spender. Am Horizont zeichnet sich aber schon die nächste Revolution ab. Durch die beschränkte Leselänge der aktuellen NGS-Technologie muss sich die Typisierung auf wenige ausgewählte, diagnostische Bereiche der HLA-Gene beschränken. Es sind allerdings Sequenziermethoden „der dritten Generation“ in Entwicklung, die diesbezüglich praktisch unlimitiert sind und die damit versprechen, völlig neue Möglichkeiten zur „Personalisierung“ des Spender/Empfänger-Abgleichs zu eröffnen.
Weitere Infos und den Artikel in der BMC Genomics finden Sie auf bmcgenomics.biomedcentral.com – alle Infos zum Labor auf dkms-lab.de
